Científicos de la Universidad de Copenhague, liderados por el español Guillermo Montoya, están investigando las características de diferentes bisturíes moleculares de CRISPR para tratar de avanzar en lo que llaman la ‘navaja suiza’ de la edición genética. El equipo ha logrado visualizar las estructuras atómicas de las proteínas Cpf1 y Cas9 y ha comprobado sus particularidades a la hora de cortar el ADN, que las hacen idóneas para diferentes usos en el corta-pega genético.
En la búsqueda de una tecnología de edición genética CRISPR multiusos, investigadores de todo el mundo están tratando de hacerla más eficaz. Para conseguirlo, han puesto el foco en las diversas proteínas que cortan de forma específica el ADN.
El equipo de Guillermo Montoya en la Novo Nordisk Foundation Center for Protein Research de la Universidad de Copenhague trabaja activamente en este campo. Recientemente, consiguió visualizar por primera vez la estructura molecular del complejo CRISPR-Cpf1, una proteína de la familia Cas que permite desenrollar el ADN para poder cortarlo e iniciar el proceso de modificación.
Esta propiedad “hará posible hacer cambios y editar las instrucciones contenidas en el genoma de manera más segura, debido a que Cpf1 reconoce la secuencia apropiada del ADN con mayor precisión”, explica Montoya a Sinc.
Ahora, en un trabajo publicado en Nature Structural & Molecular Biology los científicos de la universidad danesa han comparado el funcionamiento de estas tijeras moleculares con el de la proteína Cas9, una herramienta CRISPR de corta-pega genético con la que se puede editar de forma sencilla y barata el ADN de cualquier organismo, y que fue descubierta por las investigadoras Jennifer Doudna y Emmanuelle Charpentier en 2012.
CRISPR Cas9 ya está siendo usada para modificar genes de animales y plantas. También en terapias humanas como el cáncer y las enfermedades de retina, y sus aplicaciones no paran de crecer.
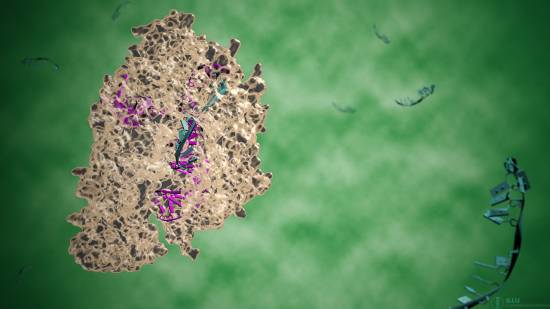
Ilustración del complejo CRISPR-Cpf1. Guiada por una molécula de ARN, la proteína Cpf1 puede ser programada para reconocer y cortar una secuencia específica en el genoma. / llusciences
Cristalografía de rayos X
Mediante una técnica de cristalografía de rayos X, Montoya y sus colegas han sido capaces de visualizar las estructuras atómicas de Cpf1 y Cas9 para entender mejor cómo interactúan con la secuencia de ADN complementaria y la manera en la que cortan la molécula de ADN.
La principal conclusión, según el biólogo molecular español, es que “dependiendo de cómo se quiera actuar sobre el ADN: bien reparar, bien inactivar o insertar un segmento de ADN en una región del genoma, algunas de estas herramientas podrán ser más apropiadas que otras debido a sus características moleculares”.
Montoya detalla que “al cortar el ADN, Cas9 genera extremos romos, por lo que esta proteína puede ser más apropiada para inactivar un gen. En cambio, Cpf1 produce extremos complementarios, que la hace más adecuada para insertar un segmento de ADN”.
En su opinión, “desvelar en detalle el engranaje de estos complejos bisturíes moleculares es esencial no solo para comprender su modo de acción, sino también para diseñar herramientas de modificación genética más eficaces y seguras que puedan ser aplicadas en el tratamiento de enfermedades, en biotecnología y en biología sintética”, concluye el investigador.
Referencia bibliográfica:
Stefano Stella, Pablo Alcón, Guillermo Montoya. “Class 2 CRISPR–Cas RNA-guided endonucleases: the Swiss Army knives of genome editing”. Nature Structural & Molecular Biology (noviembre, 2017).