Investigadores de la Universidad Autónoma de Barcelona han encontrado evidencias genéticas de adaptaciones en 2.859 regiones del genoma, incluidas algunas bien conocidas como las responsables de la tolerancia a la leche o de la adaptación a la altitud. Los datos son fruto del proyecto PopHumanScan, un catálogo exhaustivo de regiones que muestran evidencias de la selección natural en el genoma humano.
Un estudio del grupo de investigación Bioinformática de la Diversidad Genómica de la Universidad Autónoma de Barcelona (UAB), publicado en la revista Nucleic Acids Research, incrementa en un 40% el total de las señales de selección natural en el genoma humano detectadas hasta la fecha. Los investigadores han conseguido sumar un total de 873 nuevas regiones del genoma humano como firmes candidatas de haber sido el blanco de la selección natural en el algún momento desde el surgimiento de nuestra especie hasta el presente. Estas se suman a las 1.986 que ya se habían detectado hasta la fecha, proporcionado un conjunto de datos muy valiosos para responder a la pregunta: ¿qué nos hace humanos?
La gran cantidad de datos genómicos propiciados por la revolución genómica ha cambiado drásticamente la visión evolutiva del pasado humano, retando interpretaciones y resolviendo disputas que arqueólogos, historiadores, antropólogos y lingüistas han mantenido durante tiempo. Al colonizar casi todos los ambientes terrestres, nuestra especie se ha visto sometida a continuos desafíos adaptativos. Estas presiones selectivas dejan firmas en las regiones afectadas del genoma que podemos inferir analizando la variación genética.
En 2018, el grupo de investigación Bioinformática de la Diversidad Genómica de la Universidad Autónoma de Barcelona (UAB), en colaboración con científicos del Instituto de Biología Evolutiva (IBE), publicaron PopHuman, el mayor inventario de medidas de diversidad genética computadas a lo largo del genoma humano, utilizando para ello los datos del proyecto 1000 genomas.
A partir de PopHuman, los investigadores de la UAB han escaneado un conjunto de 8 medidas, que detectan huellas de selección diferentes y abarcan escalas temporales distintas, a lo largo del genoma. La detección de estas regiones en nuestra especie nos permite valorar el impacto genómico general así como determinar las variantes genómicas específicas responsables de las distintas adaptaciones humanas.
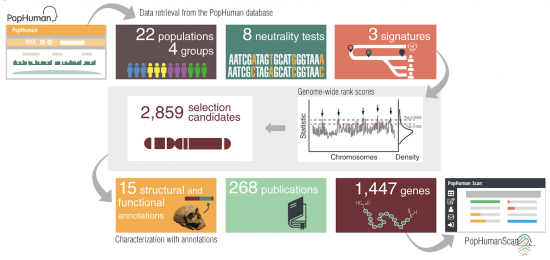
Diagrama de flujo utilizado para detectar y caracterizar estructural y funcionalmente las regiones candidatas a estar bajo selección del genoma humano
Toleracia a la lactosa, hipoxia e hibridación con neandertales
El estudio incluye información de 22 poblaciones humanas y un total de 2.859 regiones candidatas bajo selección. Un total de 1.986 de estas regiones ya habían sido detectadas previamente. El nuevo estudio de la UAB provee por lo tanto un 40% de nuevas señales genómicas relevantes en la adaptación humana, algunas relacionadas con la hibridación de nuestra especie con los neandertales y otras especies de homínidos.
Entre los resultados se incluyen ejemplos bien conocidos de adaptaciones locales humanas, como las recurrentes adaptaciones producidas en la región que contiene el gen LCT, que codifica la enzima responsable de la degradación de la lactosa.
Otro ejemplo clásico de adaptación local lo encontramos en la región que contiene el gen EGLN1, relacionado con la ruta del factor inducible por hipoxia (HIF, por sus siglas en inglés) y que estaría relacionado con la capacidad de vivir a altas altitudes, como las poblaciones Tibetanas.
Se prevé que el estudio futuro de las nuevas señales genómicas detectadas permitirá explicar nuevos ejemplos de adaptación humana, así como mejorar nuestro conocimiento acerca de cómo la introgresión de genomas arcaicos ha modelado nuestros genomas.
Los resultados se han compilado en el nuevo catálogo PopHumanScan. Se ha diseñado como una base de datos colaborativa, incluyendo por primera vez numerosas anotaciones estructurales y funcionales de las regiones, así como la recurrencia de señales de selección en las distintas poblaciones analizadas.
PopHumanScan aspira a convertirse en un repositorio central para compartir estudios de selección en humanos, guiar estudios futuros y ayudar a mejorar nuestra comprensión de cómo la selección ha modelado nuestros genomas como respuesta a los cambios en el entorno o el estilo de vida de las poblaciones humanas.
Referencian bibliográfica:
Jesús Murga-Moreno, Marta Coronado-Zamora, Alejandra Bodelón, Antonio Barbadilla, Sònia Casillas; "PopHumanScan: the online catalog of human genome adaptation", Nucleic Acids Research, Volume 47, Issue D1, 8 January 2019, Pages D1080–D1089, https://doi.org/10.1093/nar/gky959 Factor de impacto: 11.561.